细胞自噬活动是真核细胞内高度保守的生物学现象,它是在自噬相关基因的调控下利用溶酶体降解自身细胞质蛋白和受损细胞器的过程。自噬活动对细胞内环境的稳态的维持至关重要,与神经退行性疾病、肿瘤、自身免疫性疾病、代谢性疾病等多种人类重大疾病密切相关。细胞自噬是一个复杂多步骤调控过程,其晚期自噬体-溶酶体的融合是其关键步骤。主要涉及RAB蛋白、SNARE及HOPS复合体及一系列特异性调控因子等参与介导。因此,阐明自噬融合关键调控过程,对人类自噬疾病的治疗及靶向药物的研制具有重要意义。
2023年9月9日,公司细胞调控与分子药物“111”引智基地、发酵工程教育部重点实验室、教育部工业发酵省部共建协同创新中心、工业微生物湖北省重点实验室唐景峰/周策凡课题组在Nature子刊Cell Death & Differentiation(中科院TOP 1区, IF=12.4)发表了题为“RUNDC1 inhibits autolysosome formation and survival of zebrafish via clasping ATG14-STX17-SNAP29 complex ”的科研成果 (https://www.nature.com/articles/s41418-023-01215-z)。公司为成果署名第一单位,张瑞为第一作者,唐景峰/周策凡为共同通讯作者。首先,课题组利用蛋白质谱及互作技术首次发现一个新的自噬融合调节关键蛋白RUNDC1,该蛋白可与自噬核心因子ATG14结合并介导自噬融合限速过程。其次,利用脂质体融合实验及溶酶体与自噬体囊泡体外融合实验证明RUNDC1通过与STX17蛋白结合并加强其在自噬体上对ATG14-STX17-SNAP29 三元t-SNARE复合物的捆绑,从而导致融合中间体STX17-SNAP29不能有效的释放,进而抑制STX17与溶酶体表面VAMP8的互作,从而负调控自噬活性。最后,该团队在斑马鱼模型中验证了RUNDC1通过自噬进而影响斑马鱼在营养应激条件下对生存的耐受性。该研究对自噬相关疾病的治疗及靶向药物的开发提出新的开发思路。
硕士研究生杨宇妍、何超为共同第一作者。该研究得到国家自然科学基金项目(32000523、32070726、82273970、32270768、31871176) 、湖北省创新群体计划项目(2023AFA026)、湖北省重点研发计划项目(2022EHB038)、武汉市曙光计划项目(2022020801020272)资助。本项研究成果的合作单位还有英国伦敦卫生与热带医学院、英国威斯敏斯特大学、华中科技大学、重庆医科大学、陆军医科大学、长沙理工大学和加拿大阿尔伯塔大学等单位。

自噬体一旦成熟即会转运到特定的位置,需要进一步与溶酶体融合形成自噬溶酶体,在溶酶体内的酸性水解酶的作用下降解物质。自噬体和溶酶体的融合是保证细胞自噬活动正常进行的一个关键步骤。近些年的研究已发现多个参与自噬体-溶酶体融合过程的调控因子,例如SNARE蛋白、ATG8家族蛋白、HOPS复合物和ATG14蛋白等。其中,STX17蛋白作为一个关键的SNARE蛋白组分,可与ATG8家族蛋白、以及另两个SNARE蛋白SNAP29和VAMP8蛋白结合以促进自噬体和溶酶体的融合过程,但是这些蛋白相互协作实现融合的具体分子机制尚不清楚。其中,蛋白解聚回收的循环再利用是调控自噬体降解速率的关键。
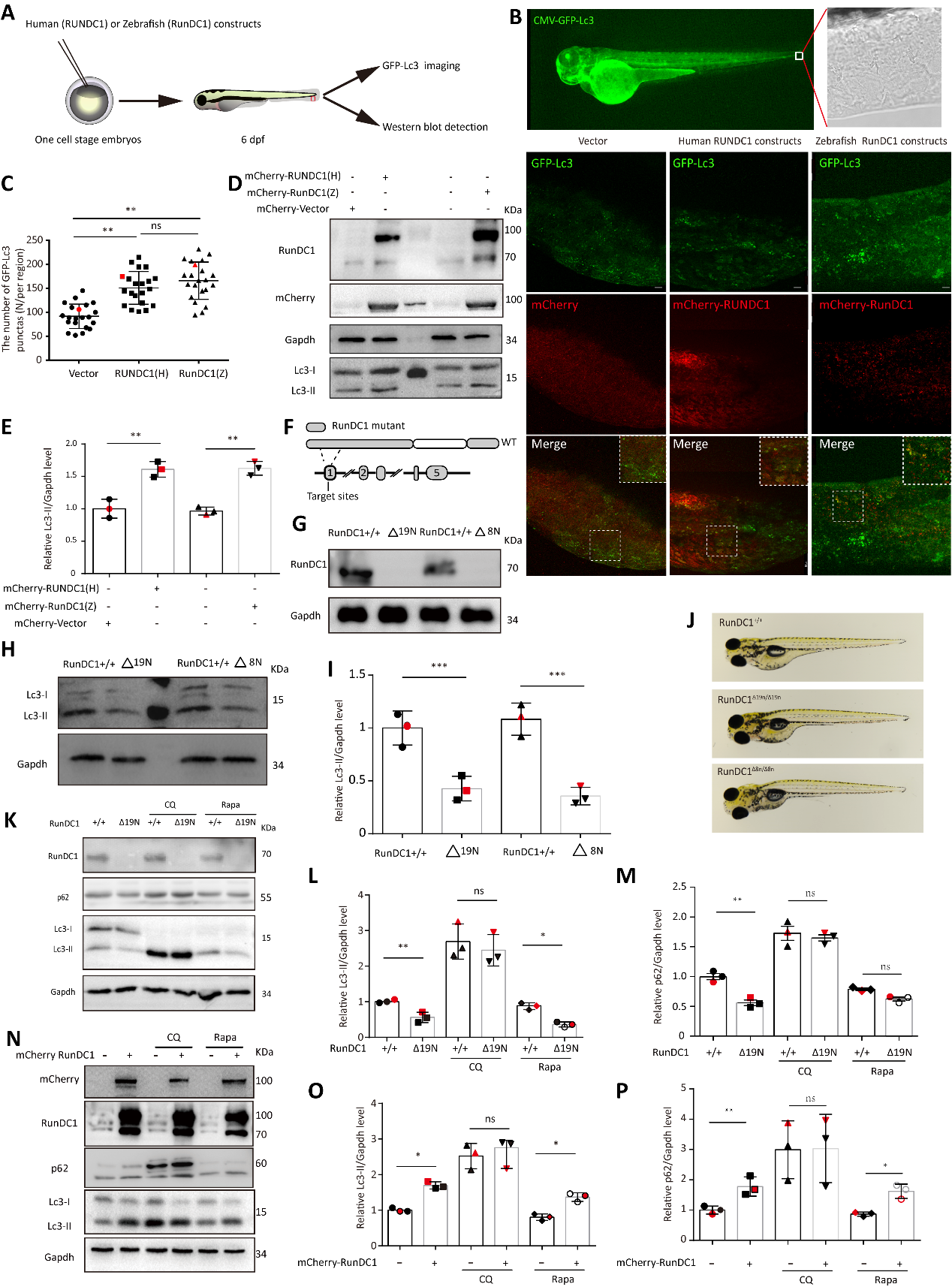
图1. RunDC1在斑马鱼模型中负调控自噬活动
这项研究中,该团队首次鉴定出与ATG14相互作用的关键融合调节蛋白RUNDC1。RUNDC1( RUN domain-containing protein 1),是一种编码613个氨基酸包含RUN结构域和螺旋结构域的蛋白质,其主要包含Coiled Coil和RUN蛋白结构域。目前,对RUNDC1的相关研究尚未见报道。
首先,该团队在细胞和Tg(CMV:EGFP-Lc3)转基因斑马鱼模型中证明了RUNDC1能够抑制自噬活性。ATG14在自噬过程中除了在自噬启动早期形成Ptdins3K-C1复合体之外,还能与UVRAG结合形成Ptdins3K-C2复合体进而调控其融合。研究者通过筛选并验证了RUNDC1与Ptdins3K-C1复合体中Beclin1、VPS34、VPS15等蛋白均不存在相互作用,而与自噬融合后期STX17、SNAP29等SNARE复合体组分结合。同时,还利用脂质体融合(lipid liposome fusion)实验及溶酶体与自噬体囊泡体外融合实验表明RUNDC1参与自噬后期融合过程。
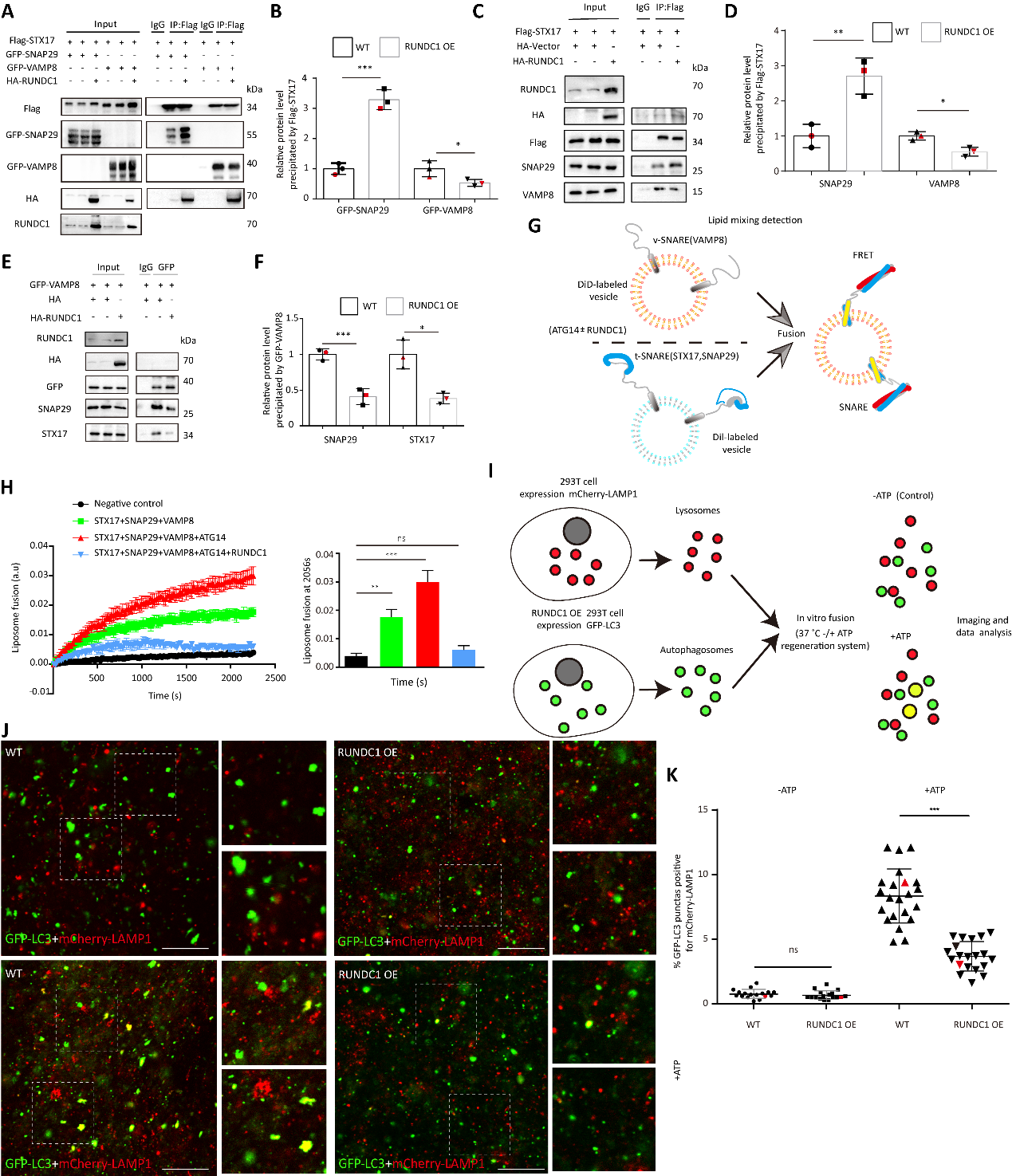
图2. RUNDC1调控STX17-SNAP29-VAMP8复合体的组装
其次,研究团队发现在自噬体上的STX17-SNAP29-ATG14复合体需要ATG14的解离才能与VAMP8 蛋白结合,进一步与溶酶体的融合。因此,该团队证明了在RUNDC1参与的情况下ATG14的同源寡聚化增强,其能够抑制ATG14从STX17-SNAP29-ATG14复合体上的解离,进而抑制了STX17-SNAP29与VAMP8的作用。即RUNDC1能够捆绑STX17-SNAP29-ATG14这一复合体,导致自噬的融合过程受阻。另外,还阐明了RUNDC1蛋白在S379位点存在磷酸化修饰,且该修饰能够促进其对自噬融合的抑制作用。

图3.RUNDC1能够调控ATG14的同源寡聚化来抑制STX17-SNAP29-VAMP8复合体
最后,该团队构建了RunDC1缺失的RunDC1Δ19n/Δ19n斑马鱼系以及过表达野生型和Run结构缺失的RunDC1ΔRun斑马鱼,发现缺失RunDC1后,斑马鱼对营养应激条件下生存能力有较大提升。相较野生型RunDC1,缺失Run结构的突变体其功能发生失活。进而利用Stx17缺失的斑马鱼证明了RunDC1对斑马鱼在营养应激下对生存的抵抗力是通过调控自噬体融合而实现的。
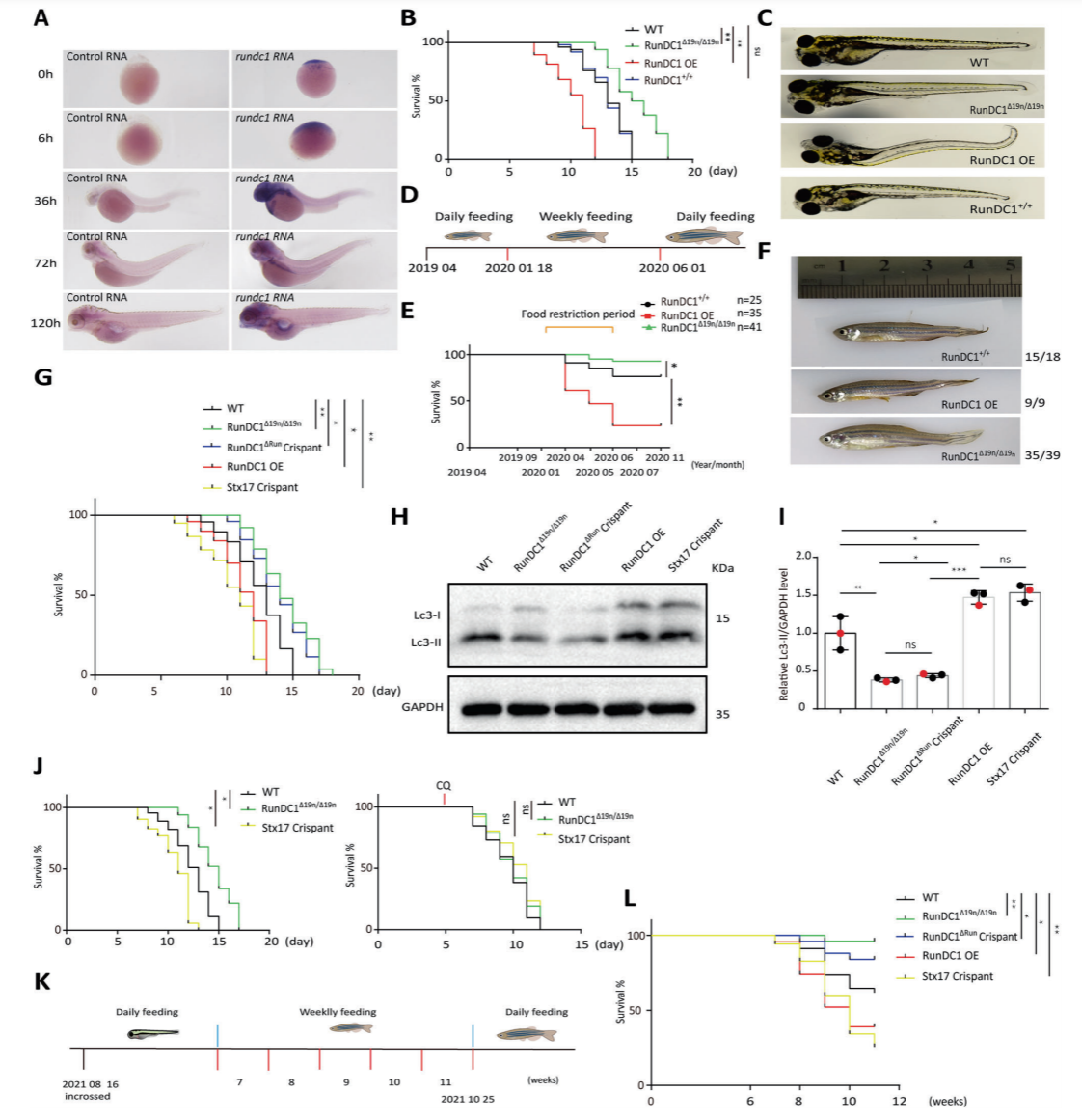
图4. RUNDC1影响斑马鱼在绝食环境下生存率
综上所述,该团队发现了一种新的自噬调控因子RUNDC1,并证明了RUNDC1定位在成熟自噬体上,阐明了其在自噬体上能够对ATG14-STX17-SNAP29 三元t -SNARE复合物进行捆绑,从而不能有效的释放融合中间体STX17-SNAP29与定位于内吞溶酶体上的VAMP8互作。同时,该研究也表明这一融合过程有可能受到了严密地调控,并且还需要其他的蛋白因子。
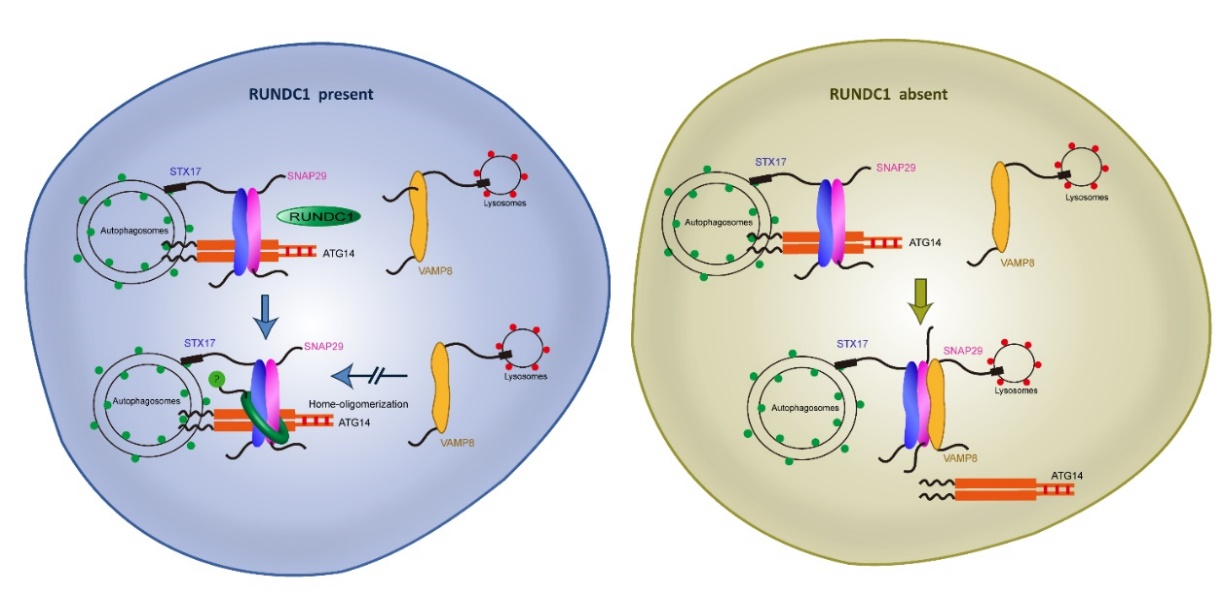
图5. RUNDC1调控STX7-SNAP29-VAMP8组装示意图
以上研究表明,自噬溶酶体形成是受到SNARE复合体调控的一个限速过程。解析自噬体融合调控的过程至关重要。本研究揭示的自噬融合限速过程的关键蛋白调节机制,将助推人类自噬失调相关疾病的新疗法发现及相关药物的研究提供新的思路。
